Top-Fragen
Zeitleiste
Chat
Kontext
Megavirus
Gattung der Familie Megaviridae Aus Wikipedia, der freien Enzyklopädie
Remove ads
Megavirus ist eine im April 2023 vom International Committee on Taxonomy of Viruses (ICTV) aufgestellte Gattung von Riesenviren in der Familie Mimiviridae.[2] Die Gattung ersetzte die vormals in der Schwestergattung Mimivirus als „Linie C“ alias „Courdo11-Gruppe“ zusammengefassten isolierten Viren und vorgeschlagenen Virusspezies. Die Gattung umfasst seitdem die Spezies Megavirus boshanense, Megavirus powaiense und Megavirus chilense; zur letzteren Art gehört der erste gefundene Vertreter der Gattung, Megavirus chilensis (MVC, MVc oder MGVC; öfters als Megavirus chiliensis verschrieben), der 2010 von französischen Wissenschaftlern vor der Küste von Chile entdeckt wurde,[4] sowie Megavirus courdo11. Zur Art Megavirus powaiense gehört Powai lake megavirus isolate 1, zu Megavirus boshanense gehört Megavirus baoshan strain SH.[5]
MGVC ist ein Verwandter von Acanthamoeba polyphaga mimivirus (APMV, Mimivirus bradfordmassiliense)[6] und infiziert ebenfalls Acanthamöben. Wegen der nur relativ entfernten Verwandtschaft[4] hat das ICTV im April 2023 die als Megavirus identifizierten Viren, die früher als Linie C vorschlagsgemäß ebenfalls der Gattung Mimivirus zugerechnet wurden, in eine eigene Gattung Megavirus (MGV) verschoben.[4][5][2]
Innerhalb der Familie der Mimiviridae wird die Gattung Megavirus zusammen mit den Gattungen Mimivirus und Moumouvirus der „Gruppe I“ zugerechnet, die die Unterfamilie Megamimivirinae inklusive Mimivirus umfasst.[2] Das ICTV ist damit im Prinzip früheren Vorschlägen wie von Schulze et al. gefolgt,[3] und diesen den Verzug gegeben gegenüber anderen Vorschlägen wie „Mimivirinae“,[7] und „Megavirinae“.[8]
Wie die Mitglieder der Gattungen Mimivirus und Moumouvirus können auch Viren der Gattung Megavirus von Virophagen befallen werden, z. B. von „Sputnik 3“ und Zamilon.[9][10][11]
Remove ads
Entdeckung
Megavirus chilensis (MVC) wurde aus Meerwasser isoliert, das im April 2010 vor der Küste von Chile in der Nähe von Las Cruces von Jean-Michel Claverie und Chantal Abergel von der Universität Aix-Marseille gesammelt wurde. Wissenschaftler aus diesem Labor waren auch an der Charakterisierung des Mimivirus ApMV beteiligt. Das Virus wurde per der Co-Kultivierung mit einer Reihe von Acanthamöben-Stämmen isoliert, und zwar von den Spezies (Acanthamoeba polyphaga, A. castellanii und A. griffini). Dabei wurde ein Verfahren benutzt, das von Timothy Rowbotham für die Isolierung intrazellulärer parasitischer Bakterien entwickelt worden war.[12] Der natürliche Wirt des MGVC ist vermutlich ein phagozytisches Protozoon, das im Meer- oder Brackwasser lebt.
Remove ads
Morphologie
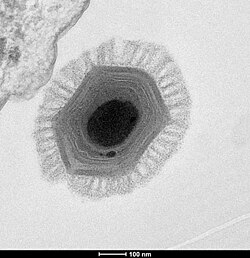
Die Virionen (Virusteilchen) des MGVC bestehen aus einem Protein-Kapsid mit einem Durchmesser von 440 nm. Sie erreichen damit fast die Ausmaße kleiner Bakterien und gehören damit zu den größten beschriebenen Viren. Die Virionen sind von einem mehrlagigen Tegument-Material von 75 bis 100 nm Schichtdicke umgeben (vgl. Mimivirus-Proteinfilamente). Das Kapsid erscheint hexagonal, allerdings ist seine ikosaedrische Symmetrie nicht perfekt, da es (mindestens ein) sogenanntes „Stargate“ enthält. Als „Stargate“ wird eine Struktur bezeichnet, die einem fünfzackigen Stern ähnelt und die eine Öffnung bildet, durch die das Kernmaterial (d. h. die Genom-DNA) des Virus in das Zytoplasma des Wirts gelangt-.[13] Das Kapsid ist von zwei Membranen umgeben, die unterschiedliche virale Proteine enthalten.
Remove ads
Genom
Zusammenfassung
Kontext
Das Genom von MGVC besteht aus einer linearen Doppelstrang-DNA mit einer Länge von 1.259.197 bp (Basenpaaren); damit war es zur Zeit seiner Entdeckung das größte bekannte Virusgenom. Es ist um 67,5 kbp größer als das Genom des Mimivirus ApMV und enthält vermutlich 1120 kodierende Regionen (i. e. kodierte Proteine), was mehr ist als bei vielen Bakterien. Der GC-Gehalt liegt bei 25 %.[14]
MGVC besitzt sieben Aminoacyl-tRNA-Synthetasen, Enzyme, die üblicherweise nur in zellulären Organismen vorkommen. Vier dieser Proteine sind auch bekannt aus Acanthamoeba polyphaga mimivirus (ApMV) und dem Acanthamoeba castellanii mamavirus (ACMV), beides Viren der Spezies Mimivirus bradfordmassiliense; nämlich diejenigen für die Aminosäuren Tyrosin, Arginin, Cystein und Methionin. MGVC enthält aber noch zusätzlich die Aminoacyl-tRNA-Synthetasen für Tryptophan, Asparagin und Isoleucin. Das Cafeteria-roenbergensis-Virus (CroV, Rheavirus sinusmexicani) enthält nur die Aminoacyl-tRNA-Synthetase für Isoleucin. Das MGVC enthält darüber hinaus das Gen für eine besondere Variante des mismatch repair Enzyms MutS, das sonst nur in den Mitochondrien von Octocorallia-Blumentiere vorkommt. Diese MutS-Version scheint nur bei Mitgliedern der Familie Mimiviridae vorzukommen.[15] MGVC enthält – ähnlich wie ApMV – Gene für den Stoffwechsel von Zuckern, Fetten und Aminosäuren.[16]
MGVC und ApMV haben 594 orthologe Gene gemeinsam. Die meisten dieser übereinstimmenden Gene befinden sich in der Mitte der viralen Genome, jeweils flankiert von artspezifischen Abschnitten im 5′- und 3′-Bereich. Bezüglich der Aminosäuresequenzen sind die durch diese orthologen Gene codierten Proteine zu etwa 50 % identisch.
Aus der Analyse des Virusgenoms von MGVC ergaben sich Hinweise darauf, dass Mimiviren und Megaviren (d. h. die Linien A und C) von einem gemeinsamen Vorfahren abstammen könnten, der durch reduktive Evolution aus Bestandteilen eines zellulären Genoms entstanden sei.[17]
Replikation
Zusammenfassung
Kontext

Die Replikationsstadien von MGVC ähneln denjenigen des Mimivirus ApMV. Kurz nach der Phagozytose und der Freisetzung des inneren Kapsids in das Zytoplasma der Wirtszelle beginnt die Eklipse-Phase. Man findet dabei sog. zytoplasmatische „Seeds“, die in ihrer Größe dem Megavirus-Kern (ohne die Hülle) entsprechen und sich innerhalb von 14 Stunden zu Virion-Fabriken entwickeln.[A. 1] Bis zur Lyse der Amöbenzelle und der Freisetzung der Viren dauert es beim MGVC gewöhnlich ca. 17 Stunden (im Gegensatz zu ca. 12 Stunden bei Mimivirus ApMV). Es werden dabei ca. 500 neue Viren pro infizierter Zelle produziert – im Gegensatz zu 1000 Partikeln bei ApMV.
MGVC gehört damit zum Phylum Nucleocytoviricota, einer Gruppe von großen zytoplasmatischen Viren, früher auch bekannt als Nucleocytoplasmic large DNA viruses (NCLDV). Als Klassifikationsmerkmal dieses Phylums gilt, dass zugehörigen Viren sich vollständig im Zytoplasma des Wirtes mithilfe sogenannter large virion factories (großer Virus-Fabriken) replizieren, ohne (wie sonst bei dsDNA-Viren üblich) ihr Genom mithilfe der wirtseigenen DNA-Polymerasen im Zellkern der Wirtszelle zu replizieren.
Remove ads
Bedeutung der Entdeckung
Zwei Eigenschaften von MGVC sind von Bedeutung: Nur 6 % größer als ApMV, so war bereits bei seiner Entdeckung anzunehmen, dass es nicht das größte Virus ist. Die Tatsache, dass drei weitere Aminoacyl-tRNA-Synthetasen in Virengenomen gefunden wurden, ließ die Vermutung aufkommen, dass diese Enzyme nicht durch einen lateralen Gentransfer von anderen Viren erworben (Virus zu Virus, setzt Doppelinfektion voraus) wurden. Die Analyse Der DNA-Sequenz zeigte dagegen Verwandtschaft mit eukaryotischen Genen an. Davon ausgehend wurde vermutet, dass das Genom dieser Viren ähnlich wie bei anderen Parasiten durch Genomreduktion aus dem Genom eines zellulären Organismus entstanden sein könnte (Wirt zu Virus, englisch host to virus, HtoV oder H2V). Weiterhin wurde vermutet, dass MGVC (bzw. die Gattung Megavirus) evolutionär sehr alt sei, und zwar eventuell älter als die heutigen Eukaryoten. Sie könnten dann gleichzeitig mit dem Zellkern entstanden sein; eine Vermutung, die durch die Ähnlichkeit des Zellkerns mit der oben erwähnten Virusfabrik unterstützt wird. Eine weitere Vermutung war, dass diese Riesenviren von einer zwischenzeitlich ausgestorbenen zellulären Domäne abstammen.[18] Diesen Ansichten wurde jedoch widersprochen und argumentiert, dass die Nucleocytoviricota (NCLDV) ihre Größe vermutlich auf mehreren Linien unabhängig durch Acquisition von Genen per horizontalem Gentransfer (H2V) erworben haben.[19] Durch die Entdeckung von Medusavirus wurde die Frage allerdings wieder neu aufgeworfen, da diese sich von anderen NCLDVs stark unterschieden, d. h. dass Gattung im Stammbaum der NCLDVs basal zu stehen schien, Das ICTV hat die Gattung Medusavirus im April 2023 dann der NCLDv-Klasse Megaviricetes ohne nähere Klassifizierung zugeordnet.[2]
Remove ads
Systematik
Zusammenfassung
Kontext




Mit der Reorganisation der Ordnung Imitervirales durch das IVTC Anfang April 2023 wurden in der Familie Mimiviridae drei Unterfamilie, darunter die Megamimivirinae eingerichtet, sowie in dieser neben der bereits bestehenden Gattung Mimivirus weitere, darunter Cotonvirus, Moumouvirus, Tupanvirus und Megavirus- Diese neuen Gattungen ersetzen auch die frühere provisorische Aufteilung der Gattung Mimivirus in Subkladen (bezeichnet als Mimivirus-Linien A, B und C). Frühere Zuordnungen von Kandidaten zur Gattung Mimivirus (wie z. B. „Mimivirus LCMiAC01“ und „02“ oder „gvSAG AB-566-O17“) sind in diesem Licht nicht mehr verbindlich, denn diese Vorschläge könnten jetzt in eine der neuen Gattungen zu klassifizieren sein.[5]
Die hier wiedergegebene Systematik basiert auf der Master Species List (MSL) #38 des International Committee on Taxonomy of Viruses (ICTV) vom 8. April 2023[2] und dem vorausgegangenen Vorschlag zur Reorganisation der Ordnung Imitervirales durch Aylward et al. (2021),[5] ergänzt um Vorschläge in doppelten Anführungszeichen nach der NCBI-Taxonomie vom 1. Mai 2023:[20]
- Unterfamilie Megamimivirinae (Vorschlag Schulz et al., 2018,[3] zu Mimiviridae Gruppe I, Mimiviren s. l., früher auch „Mimivirinae“ genannt[7])
- Gattung Megavirus (Mimiviridae: Gruppe I Linie C, MC,[21] Courdo11-Linie oder Megavirus-Linie)
- Spezies Megavirus baoshanense mit
Megavirus baoshan strain SH - Spezies Megavirus chilense[2][5] (Schreibvariante Megavirus chiliense[5]) mit
Megavirus chilensisT (Schreibvariante Megavirus chiliensisT)[22][23][24]
Megavirus lba isolate LBA111,[25][26][27]
Megavirus terra1 (kurz Terravirus 1),[28][27]
Megavirus vitis isolate vigne – isoliert aus dem Boden eines französischen Weinbergs bei Marseille,[21][29][22]
„Acanthamoeba polyphaga mimivirus“ ASM381511v1 (verschoben),
„Acanthamoeba polyphaga mimivirus“ ASM381513v1 (verschoben),
Megavirus courdo11 (kurz Courdovirus 11),[28][30][27] – vgl. auch Courdo virus CE11[31][32]
„Megavirus courdo5“ (kurz „Courdovirus 5“),[33][27]
„Megavirus courdo7“ (kurz „Courdovirus 7“),[34][30][27][23]
„Megavirus vitis transpoviron“ (mvtv)[21] - Spezies Megavirus powaiense (Powai lake megavirus, PLMV) mit
Powai lake megavirus isolate 1[30][27][35][22][36][37] - Spezies „Megavirus avenue9“[38][27]
- Spezies „Megavirus balcon“[39][27]
- Spezies „Megavirus battle43“[40][27]
- Spezies „Megavirus bus“[41][27] (veraltet: Mimivirus bus[42][43])
- Spezies „Megavirus feuillage“[44]
- Spezies: „Megavirus J3“[45][27]
- Spezies „Megavirus mammoth“[46]
- Spezies „Megavirus mont1“[47][28]
- Spezies: „Megavirus montpellier“[48](alias „Megavirus montpellier3“[27])
- Spezies: „Megavirus potager“[49]
- Spezies: „Megavirus ursino“[50][30][27]
- Spezies: „Megavirus shan“ (alias „Shan-Virus“)[51][26][27]
- Spezies: „Megavirus T1“[52][27]
- Spezies: „Megavirus T4“[53][27]
- Spezies: „Megavirus T6“[54][27]
- ?Spezies: „Megavirus caiporensis“[55]
- ?Spezies: „Bandra megavirus“ (BMV)[56][35]
- ?Spezies: „Afrovirus urmite69“[57][10]
- ohne Spezieszuweisung:
Megavirus musashi – erstmals gefunden in der Präfektur Saitama, Japan; infiziert Acanthamoeba castellanii, nicht aber A. comandoni, A. culbertsoni oder Vermamoeba vermiformis .[58]
- Spezies Megavirus baoshanense mit
- Gattung Megavirus (Mimiviridae: Gruppe I Linie C, MC,[21] Courdo11-Linie oder Megavirus-Linie)
T Bei Namensgleichheit mit der Spezies deutet ein hochgestelltes ‚T‘ einen Typus- oder Referenzstamm an.
Die Phylogenie der Mimiviridae-Gruppe I, d. h. der Unterfamilie Megamimivirinae inkl. der Gattung Mimivirus (Mimiviren s. l.) ist nach Aylward et al. (2021) vereinfacht wie folgt:[5]
| Mimiviridae-Gruppe I |
| ||||||||||||||||||||||||||||||||||||||||||
Frühere Analysen wie die von Abrahão et al. (2018) hatten für die innere Phylogenie der Gattung Megavirus noch ein etwas anderes Bild abgegeben als es der aktuellen Aufteilung der Stämme auf die drei offiziellen Megavirus-Spezies entspricht.[42]
Remove ads
Literatur
- James L. Van Etten: Giant Viruses. In: American Scientist. Band 99, Nr. 4, 2011, S. 304, doi:10.1511/2011.91.304.
- Elodie Ghedin, Jean-Michel Claverie: Mimivirus relatives in the Sargasso sea. In: Virology Journal. Band 2, 2005, S. 62, doi:10.1186/1743-422X-2-62, PMID 16105173, PMC 1215527 (freier Volltext).
- Adam Monier, Jean-Michel Claverie, Hiroyuki Ogata: Taxonomic distribution of large DNA viruses in the sea. In: Genome Biology. Band 9, Nr. 7, 2008, S. R106, doi:10.1186/gb-2008-9-7-r106, PMID 18598358, PMC 2530865 (freier Volltext).
Remove ads
Weblinks
- Welcome to the Giant Virus site, home of mimivirus and other large DNA viruses. GiantVirus.org – eine Informationsquelle über das Genom von Riesenviren.
- Stuart Siddell: Why virus taxonomy is important, Microbiology Society, 13. Februar 2018
- Jean-Luc Goudet: Nouméavirus, un étonnant virus géant qui agit à distance. Futura Santé, 1. Mai 2017 (mit bildlichen Darstellungen von Megavirus, französisch)
Remove ads
Anmerkungen
- Die zytoplasmatischen Seeds sind daher Vorstufen der Virus-Fabriken und enthalten nicht viel mehr als das Virus-Genom.
Einzelnachweise
Wikiwand - on
Seamless Wikipedia browsing. On steroids.
Remove ads