トップQs
タイムライン
チャット
視点
複製起点
ゲノムの複製が開始されるゲノム上の特定の配列 ウィキペディアから
Remove ads
複製起点または複製開始点、レプリケーター(ふくせいきてん/かいしてん、英: origin of replication, replication origin, replicator)は、ゲノムの複製が開始される、ゲノム上の特定の配列である[1]。遺伝物質が世代間で伝達されるためには、細胞分裂に先立ってDNAが半保存的複製によって適切な時期に正確に複製され、各娘細胞が染色体を全て受け取ることが必要である[2]。この過程は原核生物や真核生物などの生物ではDNAの複製、ウイルスの場合はDNAまたはRNA(二本鎖RNAウイルスなどの場合)の複製を伴う[3]。娘鎖の合成は複製起点と呼ばれる非連続的な特定の地点から始まり、全てのゲノムDNAが複製されるまで双方向的に進行する。こうしたイベントの基本的性質は共通であるものの、生物は多様な複製開始の制御戦略を進化させている[2]。
Remove ads
歴史
19世紀後半のグレゴール・メンデルによるエンドウの形質の遺伝に関する先駆的業績は、世代間の形質の移行を特定の「因子」(今日では遺伝子として確立されている)が担っていることを示唆していた[4]。当初はタンパク質が遺伝物質として機能すると推測されていたが、一世紀の後にアベリー、マクロード、マッカーティは、フリードリッヒ・ミーシェルによって発見されていたDNAが遺伝情報を運んでいることを確立した[5]。こうした発見はDNAの化学的性質や遺伝情報のコーディングの法則を解明する研究への道を開き、最終的にはワトソンとクリックによるDNAの二重らせん構造の提唱がもたらされた[6]。このDNAの三次元モデルは細胞分裂に先立って遺伝情報が半保存的に複製される機構の可能性を示しており、後にその仮説はメセルソンとスタールによる、親鎖と新生DNA鎖を区別するために同位体の取り込みを利用した実験で支持された[7][8]。その後、コーンバーグらによって新たなDNA鎖の合成を触媒する酵素であるDNAポリメラーゼが単離されたことで、生物学的なDNA複製機構のさまざまな構成要素が、まずは細菌のモデル生物である大腸菌Escherichia coliで、そして後には真核生物でも同定された[2][9]。
Remove ads
特徴
DNA複製の必要条件として重要なのは、細胞周期中で正確に1度だけ、非常に高い正確性と効率で行われることであり、それによって細胞や生物の生存に悪影響を及ぼす可能性のある遺伝的変化の蓄積が防がれる[10]。不完全で、エラーが多く、不適切な時期に行われるDNA複製は、突然変異、染色体の倍数性や異数性、遺伝子のコピー数の変化を引き起こす場合があり、これらはがんなどの疾患の原因となる場合がある[11][12]。ゲノム全体の完全かつ正確な複製と子孫細胞への遺伝情報の適切な流れを保証するため、全てのDNA複製のイベントは細胞周期の指示によって緊密に調節されているだけでなく、転写やDNA修復など他のイベントと協調して行われている[2][13][14][15]。さらに、複製起点の配列は全ての生物界を通じて一般的にAT含量が高い。これはアデニンとチミンの反復はグアニンとシトシンのリピートほど塩基のスタッキング相互作用が強固でなく、DNA鎖の分離が容易なためである[16]。
DNA複製はいくつかの段階に分割される。開始段階では、レプリソームと呼ばれる複製装置がDNA上に双方向へ向けて組み立てられる。こうした組み立てが行われる部位がDNA複製の開始部位であり、複製起点である。伸長段階では、レプリソームは複製フォークとともに各方向へ移動してDNA二重らせんを巻き戻し、双方の親鎖を鋳型として相補的な娘DNA鎖を合成する。複製が完了すると、終結イベントによってレプリソームは解体される。細胞分裂の前にゲノム全体が複製されている限り、複製開始部位の位置がどこであろうと問題にはならないが、多くの生物はゲノム上の特定の領域を選択的に複製起点として利用していることが示されている[17][18]。複製起点の位置の制御は、同じクロマチン鋳型に作用する他の過程とDNA複製とを協調させ、DNA鎖の切断やDNA損傷を避けるために必要であると考えられている[2][12][15][19][20][21][22][23]。
Remove ads
レプリコンモデル
要約
視点
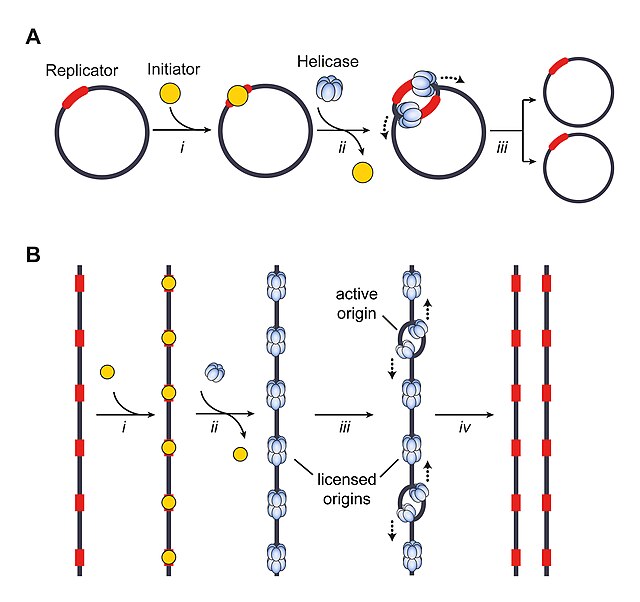
ジャコブ、ブレナー、Cuzinは大腸菌の染色体DNAの合成の調節を説明するためにレプリコン仮説を提唱した[24]。そのモデルでは、イニシエーターと呼ばれる拡散性でトランスに作用する因子が、レプリケーターと呼ばれるシスエレメントと相互作用し、複製起点近傍での複製開始を促進するとされた。イニシエーターはレプリケーターと結合すると、(多くの場合ローダータンパク質の助けを借りて)複製ヘリカーゼをDNA上に置き、その後ヘリカーゼは他のレプリソームの構成要素のリクルートと完全な複製装置の組み立てを駆動する。レプリケーターは複製開始の位置を特定し、1つの複製起点または開始イベントによって複製される染色体領域がレプリコンとして定義される[2]。
レプリコン仮説の基本的な特徴は、DNA複製の開始を制御する正の調節に依存していることであり、細菌やファージの系での多くの実験的観察を説明することができた[24]。例えば、宿主細胞へ導入された複製起点を持たない染色体外DNAは複製されないことが説明された。さらに、大腸菌で特定のプラスミドが互いの遺伝を不安定化する不和合性は、同じ分子的な開始装置を競合するためであると合理的に説明された[25]。対照的に、負の調節モデル(転写におけるオペレーターに類似したモデル)はこれらの知見を説明することができなかった[24]。しかし、ジャコブ、ブレナー、Cuzinによるレプリコンモデルの提唱後の研究では、細菌や真核生物において正負双方の調節要素からなる複製制御の多くの階層が発見され、DNA複製を時間的・空間的に制限することの複雑さと重要性が浮き彫りとなった[2][26][27][28]。
遺伝的実体としてのレプリケーターの概念は、原核生物のレプリケーター配列やイニシエータータンパク質の同定の際には非常に有用であり、また真核生物の場合でもある程度そうであったが、その構成と複雑性は生命のドメイン間で大きく異なっていた[29][30]。細菌のゲノムには通常、コンセンサスDNA配列によって特定される単一のレプリケーターが存在し、それが染色体全体の複製を制御するが、出芽酵母を除くほとんどの真核生物のレプリケーターはDNA配列のレベルでは定義されておらず、局所的なDNA構造やクロマチンの指示の組み合わせによって規定されているようである[31][32][33][34][35][36][37][38][39][40]。真核生物の染色体は細菌の染色体に比べてはるかに大きく、ゲノム全体を適切な時期に複製するためには、多くの複製起点から同時にDNA合成を開始する必要がある。さらに、特定の細胞周期での複製開始のために活性化される複製ヘリカーゼよりも多くのヘリカーゼがDNAへロードされている。文脈依存的なレプリケーターの定義や複製起点の選択が行われることは、真核生物の系におけるDNA複製プログラムは柔軟で緩やかなレプリコンモデルであることを示唆している[29]。レプリケーターと複製起点は染色体上で物理的に離れていることもあるが、多くの場合は共局在したり、近接して配置されたりしている。そのため、この項目では双方のエレメントを「複製起点」として扱う。以上をまとめると、さまざまな生物での複製起点配列の発見と単離は、複製開始機構の理解に向けた重要な出来事であった。さらに、これらの成果は細菌、酵母、哺乳類細胞内で増殖可能なシャトルベクターの開発というバイオテクノロジーにおける重要な意味を持つものでもあった[2][41][42][43]。
細菌
要約
視点

ほとんどの細菌の染色体は環状であり、単一の複製起点(oriC)を持つ。細菌のoriC領域のサイズ(250 bpから2 kbp)、配列、構成は驚くほど多様であるが[45][46]、一般的に複製の開始を駆動する能力は、細菌のイニシエーターである、DnaAと呼ばれるタンパク質によるコンセンサスDNAエレメントの配列特異的な読み出しに依存している[47][48][49][50]。細菌の複製起点には分節化されていないものと2分節されたものがあり、複製起点の活性を制御する3つの機能的エレメントが含まれる。DnaAによって特異的に認識される保存されたDNAの反復配列(DnaA-boxと呼ばれる)、ATに富むDNA unwinding element(DUE)、そして複製開始の調節を補助するタンパク質の結合部位である[17][51][52]。DnaAと二本鎖のDnaA-box領域、DUEの一本鎖DNAの双方との相互作用は複製起点の活性化に重要である。これらの相互作用はイニシエータータンパク質内の異なるドメインによって媒介されており、DnaA-boxとの相互作用はヘリックスターンヘリックス(HTH)DNA結合エレメント、DUEの一本鎖DNAとの相互作用はAAA+(ATPase associated with various cellular activities)ドメインによってそれぞれ行われる[53][54][55][56][57][58][59]。複製起点と関係したDna-boxの配列、数、配置は細菌界の種によって異なるが、それぞれの種においてこれらが特定の配置と間隔で位置していることはoriCの機能と生産的な開始複合体形成の重要である[2][45][46][60][61][62][63][64]。
細菌の中でも、大腸菌は複製起点の構成、認識、活性化の機構の研究にとって特に強力なモデル系である。大腸菌のoriCは約260 bpの領域で、DnaAに対する親和性や補因子であるATPへの依存性が異なる4つのタイプのイニシエーター結合部位が存在する。DnaA-boxのR1、R2、R4部位は高親和性部位であり、DnaAのヌクレオチド結合状態に関係なくHTHドメインが結合する[47][65][66][67][68][69]。対照的に、R部位の間に位置するI、τ、C部位は低親和性DnaA-boxであり、ATP結合型のDnaAが選択的に結合するが、特定の条件下ではADP結合型DnaAによって代替される場合もある[63][70][71][72]。高親和性・低親和性DnaA認識エレメントに対するHTHドメインの結合は、DnaA AAA+モジュールのATP依存的な高次オリゴマー化を促進する。DnaA AAA+モジュールは二本鎖DNAの外側を巻く右巻きフィラメントを形成し、超らせんのねじれを生み出すことで隣接するATに富むDUEの融解を促進する[53][73][74][75]。DNA鎖の分離は、DUEの近位領域に位置するDnaA-trioと呼ばれるトリプレットリピートがDnaA AAA+モジュールと直接相互作用することによってさらに促進される[76]。一本鎖の3ヌクレオチドがイニシエーターフィラメントと結合することでDNA鎖は伸びた構造となり、再アニーリングが防がれて開始バブルが安定化する[57]。DnaA-trioエレメントは多くの細菌種で保存されており、複製起点の機能に重要なエレメントであることが示唆される[76]。融解後のDUEは大腸菌の複製ヘリカーゼDnaBの進入部位となり、ローダータンパク質DnaCによってDNAの各一本鎖にロードされる[2]。
DnaAのさまざまなDNA結合活性は生化学的に広く研究され、さまざまなアポ型、一本鎖DNA結合型、二本鎖DNA結合型の構造が決定されているが[56][57][58][74]、複製開始時の高次のDnaA-oriCの組み立ての正確な構造は不明である。これまでに、必要不可欠なの複製起点エレメントの構成とDnaAを介したoriCの融解を説明する2つのモデルが提唱されている。2状態モデルでは、連続したDnaAフィラメントがDUEにおいて二本鎖DNA結合モード(組織化複合体)から一本鎖DNA結合モード(融解複合体)に切り替わることが想定されている[74][77]。一方、ループバックモデルでは、DNAはoriCで急激に屈曲し、イニシエーターフィラメントへ折り返されることで、DnaAプロトマーが二本鎖と一本鎖の双方のDNA領域に同時に結合するとされる[78]。oriCのDNAがDnaAによってどのように組織化されるかの解明は、今後の重要な課題である。開始複合体の構造に関する知見は、複製起点のDNAがどのように融解するかだけでなく、巻き戻されたDUEの中で露出した一本鎖DNAに対して複製ヘリカーゼがどのようにして方向性を持ってロードされるか、また、ヘリカーゼがイニシエーターや特異的ローダータンパク質とどのように相互作用してこれらのイベントを補助しているかの説明に役立つと考えられる[2]。
Remove ads
古細菌
要約
視点

古細菌の複製起点は、細菌のoriCの構成の特徴の全てではないもののその一部が共通している。細菌とは異なり、古細菌は各染色体につき複数の起点から複製を開始することが多い(1つから4つという例が報告されている)[46][79][80][81][82][83][84][85][86]。古細菌の複製起点にも、その機能を制御する特殊な配列領域が存在する[87][88][89]。こうしたエレメントには、DNA配列特異的なorigin recognition box(ORBまたはminiORB)、そして1つまたはいくつかのORB領域に隣接するATに富むDUEの双方が含まれる[85][90]。ORBエレメントは、古細菌の種間、また同じ種の複製起点の間でも、その数、配置、配列の点でかなりの多様性がみられる[80][85][91]。古細菌では、イニシエーターとしてORB領域に結合するOrc1/Cdc6によってさらなる複雑性がもたらされている。一般的に、古細菌のゲノムにはOrc1/Cdc6の複数のパラログがコードされており、これらは個々のORBエレメントに対する親和性が大きく異なり、複製起点の活性に対する寄与が異なる[85][92][93][94]。例えば、Sulfolobus solfataricusでは、染色体に3つの複製起点(oriC1、oriC2、oriC3)がマッピングされており、生化学的研究によってこれらの部位でのイニシエーターの複雑な結合パターンが明らかにされている[85][86][95][96]。oriC1に対する正しいイニシエーターはOrc1-1であり、この起点のいくつかのORBに結合する[85][93]。oriC2とoriC3にはOrc1-1とOrc1-3の双方が結合する[85][93][96]。逆に、3つ目のパラログOrc1-2は3つの複製起点全てに結合しうるが、複製開始を負に調節していると考えられている[85][96]。さらに、近縁種であるSulfolobus islandicusでは、Orc1/Cdc6とは無関係なイニシエーターであるWhiPタンパク質が全ての複製起点に結合し、oriC3の活性を駆動することが示されている[93][95]。古細菌の複製起点はいくつかのORBエレメントが近接していることが多いため、Orc1/Cdc6の複数のパラログが同時に複製起点へリクルートされ、オリゴマー化する場合もある[94][97]。しかし、細菌のDnaAとは対照的に、古細菌ではイニシエーターの高次構造への組み立ては複製起点の機能の一般的な必要条件とはなっていないようである[2]。
構造生物学的研究により、古細菌のOrc1/Cdc6がどのようにORBエレメントを認識し、複製起点のDNAをリモデリングするかに関する知見が得られている[97][98]。Orc1/Cdc6パラログは2つのドメインからなるタンパク質で、AAA+ ATPアーゼモジュールがC末端のウィングドヘリックスフォールドに結合している[99][100][101]。Orc1/Cdc6とDNAの複合体構造からは、ORBエレメント内には逆向き反復配列が存在するにもかかわらず、ORBにはOrc1/Cdc6単量体が結合することが明らかにされている[97][98]。ATPアーゼ領域とウィングドヘリックス領域の双方が二本鎖DNAと相互作用するが、ORBの回文反復配列に対して非対称的な接触を行うため、Orc1/Cdc6は反復配列に対して特定の向きで結合することとなる[97][98]。興味深いことに、DUEの両側に隣接するORBまたはminiORBエレメントは各々逆向きの方向性を持っていることが多く[80][85][94][102][103]、Orc1/Cdc6のAAA+のlidサブドメインとウィングドヘリックスドメインはDUEの両側に向かい合うように配置されることが予測される[97][98]。Orc1/Cdc6の双方の領域がMCM複製ヘリカーゼと結合するため[104][105]、このORBエレメントとOrc1/Cdc6の特異的な配置は2つのMCM複合体をDUEに対称的にロードするために重要であると考えられる[85]。このようにORBのDNA配列がOrc1/Cdc6の結合の方向性を決定する一方で、このイニシエーターはDNAとの配列特異的な相互作用は比較的少ない[97][98]。しかしながら、Orc1/Cdc6はDNAを大きく巻き戻して屈曲させることから、複製起点の認識はDNAの配列と文脈依存的な構造的特徴の双方の組み合わせに依存していることが示唆される[97][98][106]。結晶構造中ではOrc1/Cdc6の結合に伴って歪んだ二本鎖DNAでも塩基対形成は維持されている一方で、生化学的研究では古細菌のイニシエーターが細菌のDnaAと同様にDNAを融解できるかどうかに関しては矛盾する結果が得られている[93][94][107]。古細菌と真核生物のイニシエーターや複製ヘリカーゼの進化的関係からは、古細菌のMCMは二本鎖DNAに対してロードされている可能性が高いと考えられるが(次節参照)、古細菌の系での複製起点の融解とヘリカーゼのローディングの時間的な順序や、複製起点のDNA融解の機構はまだ明確には確立されていない。同様に、MCMヘリカーゼがどのようにDNAにロードされるのかに関しても、今後の研究で明らかにされるべき課題である[2]。
Remove ads
真核生物
要約
視点

真核生物における複製起点の構成や指定、そしてその活性化は細菌や古細菌のものよりもはるかに複雑であり、原核生物で確立された複製開始のパラダイムとは大きく異なっている。真核生物細胞のゲノムサイズは大きく(出芽酵母Saccharomyces cerevisiaeでは12 Mbp、ヒトでは3 Gbp)、各細胞周期の間に全ての染色体のDNA複製を完了するためには数百か所(出芽酵母)から数万か所(ヒト)の複製起点からDNA複製を開始する必要がある[27][36]。出芽酵母と関連するサッカロミケス亜門Saccharomycotinaの種を除いて、真核生物の複製起点にはコンセンサス配列は存在しないが、それらの位置はDNAの局所的なトポロジー、構造的特徴、クロマチン環境などの文脈からの指示の影響を受ける[29][35][37]。一方で、真核生物の複製起点の機能は細胞周期のM期終盤からG1期にかけてDNAへ複製ヘリカーゼをロードする、保存されたイニシエータータンパク質にやはり依存しており、この過程は複製起点のライセンス化(licensing)と呼ばれる[110]。細菌のものとは異なり、真核生物の複製ヘリカーゼは不活性な二重六量体型として複製起点の二本鎖DNAへロードされ、それらの一部のみ(哺乳類細胞では10%から20%)がS期に活性化される。この過程は複製起点の発火(firing)と呼ばれる[111][112][113]。そのため、真核生物では活性型の複製起点は、可能性のある起点全てに印をつけるライセンス化と、複製装置の組み立とDNA合成の開始が可能となる起点を選択する発火という、少なくとも2つの異なるレベルで決定される。ライセンス化された余剰の複製起点はバックアップとして機能し、近接する複製フォークの進行が遅くなったり停止したりした場合に活性化され、細胞が複製ストレスに遭遇した場合でもDNA複製が完了されるよう保証している[114][115]。ストレスが存在しない場合には、余剰の複製起点の発火は複製と関係したシグナル伝達機構によって抑制される[116][117]。このように、余剰のライセンス化複製起点の存在と、細胞周期における複製起点のライセンス化と発火の厳密な制御は、複製の過不足を防ぎ、真核生物のゲノムの完全性を維持するための2つの重要な戦略となっている[2]。
出芽酵母での初期の研究では、真核生物の複製起点も原核生物のもののようにDNA配列特異的に認識されている可能性が示されていた。出芽酵母では、レプリケーターの探索により、染色体外DNAでの効率的なDNA複製開始を補助するARS(autonomously replicating sequences)が同定された[118][119][120]。こうしたARS領域は約100–200 bpの長さで、分節化された構成をしており、A、B1、B2、そして時によりB3と呼ばれるエレメントがともに複製起点の機能に必要不可欠な役割を果たしている[121][122]。Aエレメントは保存された11 bpのACS(ARS consensus sequence)を含み[123][124]、B1エレメントとともに、真核生物の複製イニシエーターであるヘテロ六量体型複製起点認識複合体(ORC)の主結合部位を構成する[125][126][127][128]。ORCは、5つのサブユニットが保存されたAAA+ ATPアーゼとウィングドヘリックスフォールドを持ち、DNAを囲む五量体リングへと組み立てられると考えられている[128][129][130]。出芽酵母のORCでは、ATPアーゼドメインとウィングドヘリックスドメインのDNA結合エレメントは、ORCサブユニットの一部に存在する塩基性パッチ領域とともに、ORCリングの中心部のポアに配置され、ATP依存的にACSの配列特異的認識を補助する[128][131]。対照的に、B2エレメントとB3エレメントの役割は明確にはされていない。B2領域の配列はACSと類似しており、特定の条件下で2番目のORC結合部位となるか、または複製ヘリカーゼのコアの結合部位となることが示唆されている[132][133][134][135][136]。B3エレメントは転写因子Abf1をリクルートするが、B3は出芽酵母の全ての複製起点に存在するわけではなく、Abf1の結合も複製起点の機能に必要不可欠ではないようである[2][121][137][138]。
出芽酵母やその近縁種以外の真核生物における複製起点の認識は、複製起点の保存されたDNAエレメントの配列特異的な読み出しという形では行われていない。より一般的に真核生物種における染色体のレプリケーター配列を単離する試みは、遺伝学的手法とイニシエーターの結合や複製開始部位のゲノムワイドマッピングによる手法のいずれにおいても、複製起点の明確なコンセンサス配列の同定には成功していない[139][140][141][142][143][144][145][146][147][148][149][150]。そのため、出芽酵母における配列特異的なDNA-イニシエーター間相互作用は、真核生物における複製起点の特定の典型的な様式ではなく、この系の特殊な様式を表しているものであると考えられる。しかしながら、DNA複製は非連続的なの特定の部位から開始され、それらは真核生物のゲノム上にランダムに分布しているわけではないことから、染色体上の複製起点の位置を決定する代替的手法が存在すると考えられる。こうした機構は、DNAのアクセス性、ヌクレオチド配列の偏り(ATに富む配列とCpGアイランドの双方が複製起点と関連付けられている)、ヌクレオソームの配置、エピジェネティックな特徴、DNAのトポロジー、DNAの構造的特徴(G4モチーフなど)の間の複雑な連携や、調節タンパク質や転写による干渉などが関与する[17][18][34][35][37][151][152][144][153]。また、複製起点の性質は生物種間や種内の複製起点間でも異なるだけでなく、一部は発生や細胞分化の過程でも変化する。ショウジョウバエDrosophilaの毛包細胞のchorion遺伝子座は、複製開始の空間的かつ発生過程での制御の確立された例である。この領域は卵形成の特定の段階でDNA複製依存的な遺伝子増幅が行われる。その過程は複製起点の適切な時期かつ特異的な活性化に依存しており、複製起点特異的なシスエレメントと、Myb複合体、E2F1、E2F2などいくつかのタンパク質因子によって調節される[154][155][156][157][158]。このように、後生動物の複製起点は多くの要素の組み合わせによって指定され、多くの因子によって調節されていることから、より一般的に真核生物全体における複製開始部位の位置を決定する、統一的な特徴を同定することは困難であった[2]。
複製開始や複製起点の認識を促進するため、さまざまな種のORCは特殊な付属的ドメインを進化させている。これらは染色体上の複製起点、またはより一般的に染色体へのイニシエーターの標的化を促進すると考えられている。例えば、分裂酵母Schizosaccaromyces pombeのORCのOrc4サブユニットにはいくつかのATフックが存在し、ATに富むDNAに選択的に結合する[159]。一方、後生動物のORCでは、Orc6のTFIIB様ドメインが同様の機能を果たすと考えられている[160]。また、後生動物のOrc1にはBAHドメイン(bromo-adjacent homology domain)を持ち、H4K20me2修飾を持つヌクレオソームと相互作用する[109]。特に哺乳類細胞では、H4K20のメチル化は効率的な複製開始に必要であることが報告されており、Orc1のBAHドメインはORCの染色体への結合を促進するほか、エプスタイン-バールウイルスの複製起点依存的な複製も促進する[161][162][163][164][165]。少なくとも一部の後生動物において、これらの観察結果が機械的に関連しているかどうかは興味深い点であるが、さらなる研究が必要である。特定のDNAやエピジェネティックな特徴の認識に加えて、ORCは直接的または間接的にいくつかのパートナータンパク質(LRWD1、PHIP(DCAF14)、HMGA1aなど)と結合し、イニシエーターのリクルートを促進していると考えられる[33][166][167][168][169][170][171][172]。ショウジョウバエのORCは出芽酵母と同様にDNAを屈曲させること、またこの複合体のDNAへの結合は負の超らせんによって強化されることが報告されており、DNAの形状や展性が後生動物のゲノムへのORCの結合部位に影響を与えている可能性が示唆される[31][128][173][174][175]。ORCのDNA結合領域が、出芽酵母のような特定のDNA配列ではなく、後生動物のDNA二本鎖の構造的性質の読み出しをどのように補助しているのかについての分子的な理解には、DNAに結合した後生動物のイニシエーターの高分解能の構造情報が必要である。同様に、さまざまなエピジェネティックな因子が後生動物のイニシエーターのリクルートに寄与しているかどうか、またどのように寄与しているかについても、十分に理解されておらず、より詳細に記載されるべき重要な問題である[2]。
ORCとそのコファクターであるCdc6とCdt1は複製起点にリクルートされると、Mcm2-7複合体のDNAへの配置を駆動する[110][176]。古細菌の複製ヘリカーゼのコアと同様に、Mcm2-7はhead-to-head型の二重六量体として、複製起点のライセンス化のためにDNAへロードされる[111][112][113]。S期には、ライセンス化複製起点の一部でDbf4依存性キナーゼ(DDK)とサイクリン依存性キナーゼ(CDK)がMcm2-7のいくつかのサブユニットと他の開始因子をリン酸化し、ヘリカーゼのコアクチベーターであるCdc45とGINSのリクルート、DNAの融解、そして最終的には双方向的なレプリソームの組み立てを促進する[28][177]。酵母と後生動物の双方において、複製起点はヌクレオソームが無いか枯渇した状態であり、この性質はMcm2-7のローディングに重要である。このことは、複製起点のクロマチン状態はイニシエーターのリクルートだけでなく、ヘリカーゼのローディングも調節することを示している[145][178][179][180][181][182]。クロマチンがpremissiveな状態にあることは複製起点の活性化に重要であり、複製起点の効率と発火の時期の双方の調節と関係していることが示唆されている。ユークロマチンの複製起点は一般的に活性型のクロマチン標識を含んでおり、早期に複製され、そして一般的に抑制型の標識によって特徴づけられ、より後の時点で複製されるヘテロクロマチンの複製起点よりも高効率である[27][180][183]。いくつかのクロマチンリモデリング因子やクロマチン修飾酵素が複製起点や特定の開始因子に結合することが知られているが[184][185]、これらの活性がさまざまな複製開始イベントにどのように影響を与えているのかはほとんど明らかにされていない。また哺乳類細胞では、シスに作用するECRE(early replication control element)と呼ばれる配列が複製のタイミングの調節を補助し、ゲノムの三次元的構造に影響を与えていることが近年同定された[186]。ゲノムの三次元的な組織化、局所的および高次のクロマチン構造と複製開始の間の複雑な相互作用を調整する分子的および生化学的な機構の理解は、今後の研究の興味深い点である[2]。
後生動物の複製起点は多くの場合プロモーター領域と共局在していることがショウジョウバエや哺乳類の細胞で観察されており、また複製と転写を担う分子装置間の衝突はDNA損傷をもたらす場合があるため、転写と複製を適切に調整することはゲノムの安定性の維持に重要であると示唆される[20][21][140][142][144][147][187][188]。また近年の研究では、染色体へのMcm2-7のローディングの阻害またはロードされたMcm2-7の移動のいずれかの形で、転写が複製起点の位置に影響を与える、より直接的な役割が指摘されている[153][189]。配列非依存的な(しかし必ずしもランダムではない)イニシエーターのDNAへの結合は、ヘリカーゼのローディング部位を柔軟に指定することを可能にする。また、転写による干渉やライセンス化された複製起点の活性化効率のばらつきとともに、複製起点の位置の決定や、発生や細胞運命の転換時のDNA複製と転写プログラムの共調節に寄与していると考えられる。分裂酵母の開始イベントの計算機モデリングの結果や、後生動物における細胞種特異的かつ発生過程で調節される起点の同定は、この考えと符合している[141][149][153][190][191][192][193][194]。1つの集団内のさまざまな細胞間では複製起点の選択に大きな柔軟性が存在するが[144][150][191]、複製起点の利用の不均一性をもたらす分子機構はまだ明確になっていない。後生動物の系において1細胞での複製起点のマッピングを行い、これらの開始イベントと1細胞での遺伝子発現やクロマチン状態とを関連付けることは、複製起点の選択が純粋に確率的なものなのか、それとも明確な方法で制御されているのかを明らかにする上で重要である[2]。
Remove ads
ウイルス

ウイルスは多くの場合、単一の複製起点を持つ。
ウイルスの複製に関与するさまざまなタンパク質が記載されている。例えば、ポリオーマウイルスは宿主細胞のDNAポリメラーゼを利用する。DNAポリメラーゼはT抗原が存在する場合にウイルスの複製起点に結合する。
多様性
DNA複製は遺伝子の継承に不可欠であるが、すべての染色体が完全にコピーされて遺伝子のコピー数が維持される限り、明確な部位特異的な複製起点はゲノム複製に厳密に必要とされる条件ではない。例えば、特定のバクテリオファージやウイルスは専用の複製起点に依存せず、相同組換えによってDNA複製を開始することができる[195]。同様に、古細菌Haloferax volcaniiは、内在性の複製起点が欠失した際には組換え依存的な開始を利用してゲノムを複製する。大腸菌や出芽酵母でも、切断によって誘導されたり転写によって開始されたりする、同様の非典型的な開始イベントが報告されている[196][197][198][199][200]。このような例外的な状況下でも細胞は生存を維持できるにもかかわらず、複製起点依存的な開始は生命のさまざまなドメインで普遍的に利用されている共通した戦略である[2]。
複製開始の詳細な研究は、限られた数のモデル系に焦点を当ててきた。広く研究されている菌類や後生動物はいずれもオピストコンタのスーパーグループに属しており、真核生物の進化のほんの一部を表しているにすぎない[201]。キネトプラストやテトラヒメナなど他の真核生物のモデル系では、比較的わずかな研究しか行われていない[202][203][204][205][206][207][208]。驚くべきことにこれらの研究では、複製起点の特性とイニシエーターの構成の双方において、酵母や後生動物との興味深い違いが明らかとなっている[2]。
Remove ads
出典
関連文献
関連項目
外部リンク
Wikiwand - on
Seamless Wikipedia browsing. On steroids.
Remove ads
