상위 질문
타임라인
채팅
관점
핵산 이중 나선
핵산의 이중 가닥 분자에 의해 형성되는 구조 위키백과, 무료 백과사전
Remove ads
이중 나선[1](double helix)은 분자생물학에서 DNA와 같은 핵산의 이중 가닥 분자가 형성하는 구조이다. 핵산 복합체의 이중 나선형 구조는 2차 구조의 결과로 발생하며, 3차 구조를 결정하는 기본 요소이다. 이 구조는 로절린드 프랭클린, 그녀의 학생 레이몬드 고슬링, 그리고 동료 과학자 모리스 윌킨스, 제임스 D. 왓슨, 프랜시스 크릭에 의해 발견되었으며,[2] '이중 나선'이라는 용어는 1968년 왓슨의 더블 헬릭스: DNA 구조 발견에 대한 개인적인 이야기 출판으로 대중문화에 들어왔다.
이 문서에는 관련 분야 전문가의 참여가 필요합니다. |

핵산의 DNA 이중 나선 생체고분자는 서로 염기쌍을 이루는 뉴클레오타이드에 의해 함께 유지된다.[3] 자연에서 가장 흔한 이중 나선 구조인 B형 DNA는 1회전당 약 10~10.5개의 염기쌍을 가진 오른손잡이 이중 나선이다.[4] DNA의 이중 나선 구조는 주 홈(major groove)과 부 홈(minor groove)을 포함한다. B형 DNA에서 주 홈은 부 홈보다 넓다.[3] 주 홈과 부 홈의 폭 차이를 고려할 때, B형 DNA에 결합하는 많은 단백질은 더 넓은 주 홈을 통해 결합한다.[5]
Remove ads
역사
DNA 구조의 이중 나선 모델은 1953년 제임스 D. 왓슨과 프랜시스 크릭이 학술지 네이처에 처음 발표했으며,[6] (1954년 X,Y,Z 좌표[7]) 이는 로절린드 프랭클린과 그녀의 학생 레이몬드 고슬링이 '사진 51'로 명명된 DNA의 결정적인 X선 회절 이미지를 촬영한 연구,[8][9] 그리고 모리스 윌킨스, 알렉산더 스토크스, 허버트 윌슨의 연구에 바탕을 두었으며,[10] 어윈 샤가프의 염기쌍 화학 및 생화학 정보도 활용되었다.[11][12][13][14][15][16] 그 이전에는 이미 단백질의 2차 구조 모티프의 형태를 정확하게 특성화했던 라이너스 폴링과 그의 협력자 로버트 코리는 로절린드 프랭클린의 사진을 보지 못해 DNA가 삼중 가닥 형태를 취할 것이라고 잘못 가정했었다.[17]
DNA 구조가 이중 나선이라는 사실은 생물체에서 유전 정보가 저장되고 복사되는 염기쌍 형성 메커니즘을 밝혀냈으며, 20세기 가장 중요한 과학적 발견 중 하나로 널리 알려져 있다. 로절린드는 죽은 관계로 받지 못하였고, 크릭, 윌킨스, 왓슨이 이 발견에 기여한 공로로 1962년 노벨 생리학·의학상을 공동 수상했다.[18]
Remove ads
핵산 혼성화
혼성화(Hybridization)는 상보적 염기쌍이 결합하여 이중 나선을 형성하는 과정이다. 용융(Melting)은 이중 나선 가닥 간의 상호작용이 끊어져 두 핵산 가닥이 분리되는 과정이다. 이 결합은 약하여 가벼운 가열, 효소, 또는 기계적인 힘으로 쉽게 분리된다. 용융은 핵산의 특정 지점에서 우선적으로 발생한다.[19] T와 A가 풍부한 영역은 C와 G가 풍부한 영역보다 쉽게 용융된다. 일부 염기 단계(쌍)도 T A와 T G처럼 DNA 용융에 취약하다.[20] 이러한 기계적 특성은 많은 유전자의 시작 부분에 TATA 박스와 같은 서열을 사용하여 RNA 중합효소가 전사를 위해 DNA를 용융하는 것을 돕는 데 반영된다.[21]
중합효소 연쇄 반응(PCR)에서 사용되는 것과 같이 가벼운 가열에 의한 가닥 분리는 간단하며, 분자가 약 10,000 염기쌍(10킬로염기쌍, 또는 10kbp) 미만인 경우에 해당한다. DNA 가닥의 얽힘은 긴 부분을 분리하기 어렵게 만든다.[22] 세포는 DNA-용융 효소(헬리케이스)가 DNA 회전효소와 동시에 작동하도록 하여 이 문제를 피한다. DNA 회전효소는 한 가닥의 인산 골격을 화학적으로 절단하여 다른 가닥을 중심으로 회전할 수 있도록 한다.[23] 헬리케이스는 DNA 중합효소와 같은 서열 판독 효소의 진행을 돕기 위해 가닥을 풀린다.[24]
Remove ads
염기쌍 기하학

염기 또는 염기쌍 단계의 기하학적 구조는 이동(shift), 미끄러짐(slide), 상승(rise), 기울임(tilt), 롤(roll), 비틀림(twist)의 6가지 좌표로 특징지을 수 있다. 이 값들은 나선 축을 따라 각 핵산 분자의 모든 염기 또는 염기쌍의 이전 염기 또는 염기쌍에 대한 공간에서의 위치와 방향을 정확하게 정의한다. 함께, 이들은 분자의 나선 구조를 특성화한다. 정상적인 구조가 파괴된 DNA 또는 RNA 영역에서 이러한 값의 변화를 사용하여 이러한 파괴를 설명할 수 있다.
각 염기쌍에 대해, 그 이전 염기쌍과 관련하여 고려해야 할 다음과 같은 염기쌍 기하학이 있다:[25][26][27]
- 전단(Shear)
- 신장(Stretch)
- 엇갈림(Stagger)
- 좌굴(Buckle)
- 프로펠러(Propeller): 같은 염기쌍 내에서 한 염기가 다른 염기에 대해 회전하는 것.
- 열림(Opening)
- 이동(Shift): 염기쌍 평면에서 첫 번째 축에 수직하며, 부 홈에서 주 홈으로 향하는 축을 따라 변위하는 것.
- 미끄러짐(Slide): 염기쌍 평면에서 한 가닥에서 다른 가닥으로 향하는 축을 따라 변위하는 것.
- 상승(Rise): 나선 축을 따라 변위하는 것.
- 기울임(Tilt): 이동 축을 중심으로 회전하는 것.
- 롤(Roll): 미끄러짐 축을 중심으로 회전하는 것.
- 비틀림(Twist): 상승 축을 중심으로 회전하는 것.
- x-변위
- y-변위
- 경사(inclination)
- 팁(tip)
- 피치(pitch): 나선이 한 바퀴 완전히 회전하는 동안의 높이.
상승과 비틀림은 나선의 방향성과 피치를 결정한다. 반면에 다른 좌표는 0이 될 수 있다. 미끄러짐과 이동은 B형 DNA에서는 일반적으로 작지만, A형 DNA와 Z형 DNA에서는 상당하다. 롤과 기울임은 연속적인 염기쌍을 덜 평행하게 만들며, 일반적으로 작다.
'기울임'은 과학 문헌에서 종종 다르게 사용되어, 첫 번째 가닥 간 염기쌍 축이 나선 축에 대해 수직에서 벗어나는 정도를 나타낸다. 이는 연속적인 염기쌍 사이의 미끄러짐에 해당하며, 나선 기반 좌표에서는 '경사'로 적절하게 불린다.
나선 기하학
요약
관점
적어도 세 가지 DNA 형태가 자연에서 발견되는 것으로 알려져 있으며, A형 DNA, B형 DNA, 그리고 Z형 DNA이다. 제임스 D. 왓슨과 프랜시스 크릭이 기술한 B형은 세포에서 우세한 것으로 여겨진다.[28] 이는 폭이 23.7 Å이고, 서열 10 bp당 34 Å씩 늘어난다. 이중 나선은 오른손잡이 비틀림을 가지며, 용액에서 약 10.4–10.5 염기쌍마다 축을 중심으로 한 바퀴를 완전히 회전한다. 이러한 비틀림 빈도(나선 피치라고 함)는 각 염기가 사슬의 이웃 염기에 가하는 스태킹 힘에 크게 의존한다.
A형 DNA와 Z형 DNA는 B형 DNA와 기하학적 구조 및 크기에서 크게 다르지만, 여전히 나선 구조를 형성한다. A형은 실험실의 탈수된 DNA 샘플(예: 결정학 실험에 사용되는 샘플)과 DNA와 RNA 가닥의 혼성 쌍에서만 발생한다고 오랫동안 여겨졌지만, DNA 탈수는 생체내에서 발생하며, A형 DNA는 이제 생물학적 기능을 가지는 것으로 알려져 있다. 세포가 조절 목적으로 메틸화한 DNA 세그먼트는 Z형 구조를 취할 수 있으며, 이 경우 가닥은 A형 DNA 및 B형 DNA와 반대 방향으로 나선 축을 중심으로 회전한다. 단백질-DNA 복합체가 Z형 DNA 구조를 형성한다는 증거도 있다.
다른 형태도 가능하다. A형 DNA, B형 DNA, C형 DNA, E형 DNA,[29] L-DNA (D-DNA의 광학 이성질체형),[30] P-DNA,[31] S-DNA, Z-DNA 등이 현재까지 기술되었다.[32] 사실, 현재[update] F, Q, U, V, Y 문자만이 미래에 나타날 수 있는 새로운 DNA 구조를 설명하는 데 사용할 수 있다.[33][34] 그러나 이들 형태의 대부분은 인위적으로 만들어졌으며 자연적으로 발생하는 생물학적 시스템에서는 관찰되지 않았다. 삼중 나선 DNA 형태와 G-4중 나선 및 i-모티프와 같은 4중 나선 형태도 있다.


홈
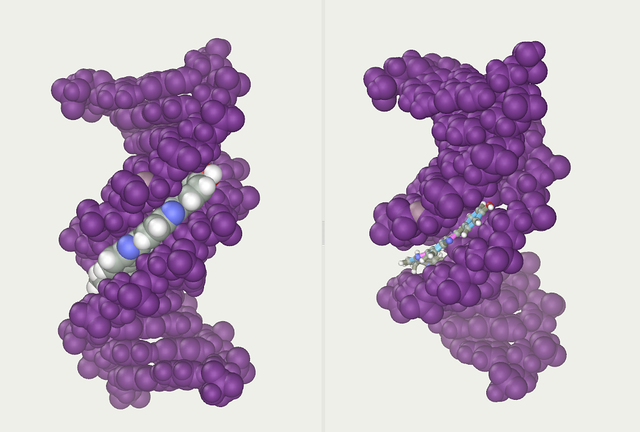
쌍을 이루는 나선 가닥이 DNA 골격을 형성한다. 가닥 사이의 공간, 즉 홈을 따라 다른 이중 나선을 찾을 수 있다. 이 빈 공간은 염기쌍에 인접하며 결합 부위를 제공할 수 있다.[38] 가닥이 서로 직접적으로 마주보지 않기 때문에 홈의 크기는 같지 않다. 한 홈인 주 홈은 폭이 22 Å이고 다른 홈인 부 홈은 폭이 12 Å이다.[39] 부 홈의 좁음은 주 홈에서 염기의 가장자리가 더 접근하기 쉽다는 것을 의미한다. 결과적으로, 이중 가닥 DNA의 특정 염기서열에 결합할 수 있는 전사인자와 같은 단백질은 일반적으로 주 홈에 노출된 염기 측면과 접촉한다.[5] 이러한 상황은 세포 내 DNA의 비정상적인 형태에서 달라지지만 (아래 참조), 주 홈과 부 홈은 DNA가 일반적인 B형으로 다시 비틀렸을 때 보이는 크기 차이를 반영하기 위해 항상 명명된다.[40]
비이중 나선 형태
비나선형 모델은 1970년대 후반에 플라스미드와 염색질의 DNA 복제 문제에 대한 잠재적인 해결책으로 잠시 고려되었다. 그러나 이후 DNA 이중체 및 뉴클레오솜 코어 입자의 엑스선결정학과 DNA 회전효소의 발견과 같은 실험적 발전으로 인해 이 모델들은 이중 나선 모델에 찬성하여 제쳐졌다. 또한, 비이중 나선 모델은 현재 주류 과학계에서 받아들여지지 않는다.[41][42]
Remove ads
굽힘
요약
관점
DNA는 비교적 단단한 고분자로, 일반적으로 웜라이크 사슬로 모델링된다. DNA는 굽힘, 비틀림, 압축의 세 가지 중요한 자유도를 가지고 있으며, 각각은 세포 내 DNA에 가능한 것에 특정 제한을 가한다. 비틀림-비틀림 강성은 DNA의 원형화와 DNA 결합 단백질의 상대적 방향에 중요하며, 굽힘-축 강성은 DNA 감싸기와 원형화 및 단백질 상호작용에 중요하다. 높은 장력이 없는 경우 압축-신장은 상대적으로 중요하지 않다.
영속 길이, 축 강성
용액 속의 DNA는 단단한 구조를 취하지 않고 열 진동과 물 분자와의 충돌로 인해 끊임없이 형태가 변하며, 이는 고전적인 강성 측정치를 적용하는 것을 불가능하게 만든다. 따라서 DNA의 굽힘 강성은 영속 길이로 측정되며, 다음과 같이 정의된다.
고분자의 굽힘 유연성은 일반적으로 영속 길이 Lp로 정량화되며, 이는 고분자가 거의 단단한 막대처럼 행동하는 길이 스케일이다. 구체적으로, Lp는 고분자의 시간 평균 방향이 상관 관계를 잃게 되는 고분자 세그먼트의 길이로 정의된다...[43]
이 값은 원자힘 현미경을 사용하여 다양한 길이의 DNA 분자를 직접 영상화함으로써 직접 측정할 수 있다. 수용액에서 평균 영속 길이는 약 50 nm(또는 150 염기쌍)로 밝혀졌다.[44] 더 넓게는 45에서 60 nm 사이[45] 또는 132–176 염기쌍(DNA 직경은 2 nm)으로 관찰되었다.[46] 이는 온도, 수용액 조건 및 DNA 길이에 따라 크게 달라질 수 있다.[45] 이로 인해 DNA는 중간 정도의 단단한 분자가 된다.[44]
DNA 부분의 영속 길이는 서열에 다소 의존하며, 이는 상당한 변화를 유발할 수 있다. 이러한 변화는 주로 염기 스태킹 에너지와 부 홈 및 주 홈으로 확장되는 잔기 때문이다.
DNA 굽힘 모델
영속 길이보다 큰 길이 스케일에서 DNA의 엔트로피 유연성은 크라트키-포로드 웜라이크 체인 모델과 같은 표준 고분자물리학 모델과 놀랍도록 일치한다.[48] 웜라이크 체인 모델과 일치하는 관찰은 DNA 굽힘이 매우 작은(피코뉴턴 미만) 힘에서 훅의 법칙에 의해 설명된다는 것이다. 영속 길이보다 짧은 DNA 세그먼트의 경우, 굽힘 힘은 거의 일정하며 동작은 웜라이크 체인 예측에서 벗어난다.
이러한 효과는 작은 DNA 분자를 쉽게 원형화하고, 고도로 굽은 DNA 부분을 찾을 확률을 높인다.[49]
굽힘 선호도
DNA 분자는 종종 굽는 데 선호하는 방향, 즉 비등방성 굽힘을 갖는다. 이것은 DNA 서열을 구성하는 염기의 특성 때문이며, 무작위 서열은 선호하는 굽힘 방향, 즉 등방성 굽힘을 갖지 않는다.
선호되는 DNA 굽힘 방향은 각 염기가 다음 염기 위에 쌓이는 안정성에 의해 결정된다. 불안정한 염기 스태킹 단계가 DNA 나선의 한쪽에만 지속적으로 발견되면 DNA는 그 방향에서 멀어지도록 우선적으로 굽을 것이다. 굽힘 각도가 증가하면 입체 장애와 잔기들이 서로에 대해 굴러가는 능력도 역할을 하며, 특히 부 홈에서 그렇다. A와 T 잔기는 굽힘 안쪽의 부 홈에 우선적으로 발견될 것이다. 이 효과는 뉴클레오솜 입자와 같이 DNA가 강하게 굽어지는 DNA-단백질 결합에서 특히 나타난다. 위에서 염기 단계 왜곡을 참조하라.
뛰어난 굽힘 선호도를 가진 DNA 분자는 본질적으로 굽어질 수 있다. 이것은 파동편모충속 키네토플라스트 DNA에서 처음 관찰되었다. 이를 유발하는 전형적인 서열은 4-6개의 T 및 A 잔기가 G 및 C가 풍부한 부분으로 분리되어 있어, 분자의 한쪽에 있는 부 홈과 A 및 T 잔기가 위상을 유지하게 한다. 예를 들면:
| ¦ | ¦ | ¦ | ¦ | ¦ | ¦ | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| G | A | T | T | C | C | C | A | A | A | A | A | T | G | T | C | A | A | A | A | A | A | T | A | G | G | C | A | A | A | A | A | A | T | G | C | C | A | A | A | A | A | A | T | C | C | C | A | A | A | C |
본질적으로 굽은 구조는 염기쌍이 서로에 대해 '프로펠러 비틀림'을 하여 염기 단계 사이에 비정상적인 이중 수소 결합이 가능하게 한다. 온도가 높아지면 이 구조는 변성되어 본질적인 굽힘이 사라진다.
비등방적으로 굽어지는 모든 DNA는 평균적으로 더 긴 영속 길이와 더 큰 축 강성을 갖는다. 이러한 강성 증가는 분자가 등방적으로 작용하게 만드는 무작위 굽힘을 방지하는 데 필요하다.
원형화
DNA의 원형화는 분자의 축(굽힘) 강성과 비틀림(회전) 강성 모두에 달려 있다. DNA 분자가 성공적으로 원형화되려면 완전히 원을 만들 수 있을 만큼 충분히 길어야 하며, 끝이 올바른 회전 방향으로 배열되어 결합이 일어날 수 있도록 올바른 수의 염기를 가져야 한다. DNA 원형화에 최적의 길이는 약 400 염기쌍(136 nm)이며, DNA 나선의 정수배 회전, 즉 10.4 염기쌍의 배수를 가져야 한다. 비정수배 회전은 원형화에 상당한 활성화 에너지 장벽을 제공한다. 예를 들어, 10.4 x 30 = 312 염기쌍 분자는 10.4 x 30.5 ≈ 317 염기쌍 분자보다 수백 배 더 빠르게 원형화될 것이다.[50]
짧은 원형화된 DNA 세그먼트의 굽힘은 균일하지 않다. 오히려, 영속 길이보다 짧은 원형화된 DNA 세그먼트의 경우, DNA 굽힘은 AT-풍부 세그먼트에서 우선적으로 형성되는 1-2개의 꼬임으로 국부화된다. 틈새가 존재하면 굽힘은 틈새 부위에 국부화될 것이다.[49]
Remove ads
신장
탄성 신장 영역
더 긴 DNA 구간은 장력 하에서 엔트로피적으로 탄력적이다. DNA가 용액 상태일 때, 용매의 열욕조에서 이용 가능한 에너지 때문에 끊임없이 구조적 변화를 겪는다. 이는 분자의 열 진동과 물 분자와의 지속적인 충돌 때문이다. 엔트로피적인 이유로, 늘어진 상태보다 더 조밀하고 이완된 상태가 열적으로 접근하기 쉽기 때문에 DNA 분자는 거의 항상 얽히고 이완된 형태로 발견된다. 이러한 이유로, 한 DNA 분자는 힘을 받으면 늘어나서 곧게 펴진다. 광 핀셋을 사용하여 DNA의 엔트로피 신장 거동이 고분자물리학적 관점에서 연구 및 분석되었으며, DNA가 생리적으로 접근 가능한 에너지 스케일에서 크라트키-포로드 웜라이크 체인 모델과 크게 유사하게 행동하는 것으로 밝혀졌다.
신장 하에서의 상전이
충분한 장력과 양의 토크 하에서, DNA는 염기들이 바깥쪽으로 퍼지고 인산기가 중앙으로 이동하는 상전이를 겪는다고 생각된다. 과신장된 DNA에 대한 이 제안된 구조는 원래 라이너스 폴링이 DNA의 가능한 구조로 제시한 것을 기리기 위해 P형 DNA라고 불렸다.[31]
강제적인 토크가 없는 상태에서 DNA의 기계적 신장에서 얻은 증거는 일반적으로 S형 DNA로 불리는 추가 구조로 이어지는 전이 또는 전이들을 시사한다. 이러한 구조는 적용된 힘 하에서 용액 내에서 원자 해상도 이미징을 수행하기 어렵기 때문에 아직 명확하게 특성화되지 않았지만, 많은 컴퓨터 시뮬레이션 연구가 수행되었다 (예:[51][52]).
제안된 S-DNA 구조에는 염기쌍 스태킹과 수소 결합을 보존하면서 기울임으로 확장을 완화하는 구조(GC-풍부)와, 염기 스택의 부분적인 용융이 일어나지만 염기-염기 결합은 전반적으로 보존되는 구조(AT-풍부)가 포함된다.
염기쌍 스택의 주기적인 파열이 3 bp당 한 번(따라서 3 bp-bp 단계 중 하나) 발생하는 규칙적인 구조로 제안되었는데, 이는 염기 스태킹의 평면성을 보존하고 적절한 양의 확장을 방출한다.[53] "Σ-DNA"라는 용어는 기억을 돕기 위해 도입되었는데, 시그마 문자의 오른쪽을 향하는 세 점이 세 개의 그룹화된 염기쌍을 상기시키는 역할을 한다. Σ 형태는 GNC 가설에 따라 진화적으로 중요하다고 여겨지는 GNC 모티프에 대한 서열 선호도를 보이는 것으로 나타났다.[54]
Remove ads
초나선 및 위상수학
요약
관점

B형 DNA 나선은 비틀림 변형이 없을 때 10.4-10.5 bp당 360° 회전한다. 그러나 많은 분자 생물학적 과정이 비틀림 변형을 유발할 수 있다. 나선 비틀림이 과도하거나 부족한 DNA 부분을 각각 양성 또는 음성 초나선이라고 한다. 생체 내 DNA는 일반적으로 음성 초나선 상태이며, 이는 RNA 전사에 필요한 이중 나선 풀림(용융)을 용이하게 한다.
세포 내 대부분의 DNA는 위상학적으로 제한된다. DNA는 일반적으로 폐쇄 루프 (예: 원핵생물의 플라스미드) 또는 확산 계수가 사실상 위상학적으로 폐쇄된 도메인을 생성하는 매우 긴 분자로 발견된다. 선형 DNA 부분도 종종 단백질이나 물리적 구조 (예: 막)에 결합하여 폐쇄된 위상학적 루프를 형성한다.
프랜시스 크릭은 DNA 초나선을 고려할 때 연결 번호의 중요성을 처음으로 제안한 사람 중 한 명이다. 1976년에 발표된 논문에서 크릭은 이 문제를 다음과 같이 설명했다.
닫힌 이중 가닥 DNA 분자에 의해 형성된 초나선을 고려할 때, 연결 번호와 비틀림과 같은 특정 수학적 개념이 필요하다. 닫힌 리본에 대한 이들의 의미와 닫힌 곡선의 꼬임 번호가 설명된다. 몇 가지 간단한 예가 제시되며, 그 중 일부는 염색질 구조와 관련이 있을 수 있다.[55]
DNA 위상학 분석은 세 가지 값을 사용한다.
- L = 연결 번호 - 한 DNA 가닥이 다른 가닥을 감싸는 횟수. 닫힌 루프의 경우 정수이며, 닫힌 위상학적 도메인의 경우 상수이다.
- T = 비틀림 - 이중 가닥 DNA 나선의 총 회전수. 이는 일반적으로 브로민화 에티듐과 같은 인터칼레이션 물질이나 DNA의 강성을 변경하는 다른 요소가 없는 경우, 위상학적으로 열린 이중 가닥 DNA 나선이 용액에서 자유롭게 만드는 회전수(염기 수/10.5)에 접근하는 경향이 있다.
- W = 꼬임 - 이중 가닥 DNA 나선이 초나선 축을 중심으로 회전하는 횟수
- L = T + W 및 ΔL = ΔT + ΔW
닫힌 위상학적 도메인에서 T의 어떤 변화도 W의 변화에 의해 균형을 이루어야 하며, 그 반대도 마찬가지이다. 이는 DNA의 고차 구조를 유발한다. 꼬임이 0인 원형 DNA 분자는 원형일 것이다. 이 분자의 비틀림이 나중에 초나선에 의해 증가하거나 감소하면 꼬임이 적절하게 변경되어 분자가 꼬임 나선형 또는 환형 초나선 꼬임을 겪게 된다.
이중 가닥 나선형 DNA 조각의 양쪽 끝이 연결되어 원을 형성하면 가닥들은 위상학적으로 매듭지어지게 된다. 이는 한 가닥을 끊지 않는 어떤 과정(예: 가열)으로도 단일 가닥을 분리할 수 없음을 의미한다. 위상학적으로 연결된 DNA 가닥을 푸는 작업은 DNA 회전효소라고 불리는 효소에 의해 수행된다. 이 효소들은 한 가닥 또는 두 가닥을 절단하여 다른 이중 가닥 또는 단일 가닥 부분이 통과할 수 있도록 함으로써 원형 DNA의 매듭을 푸는 데 특화되어 있다. 이 매듭 풀기는 원형 DNA의 복제와 유사한 위상학적 제약을 갖는 선형 DNA의 다양한 유형의 유전자 재조합에 필요하다.
연결 번호 역설
수년 동안, 진핵생물 게놈에서 잔여 초나선의 기원은 불분명했다. 이 위상학적 퍼즐은 일부에게 "연결 번호 역설"로 불렸다.[56] 그러나 뉴클레오솜의 실험적으로 결정된 구조에서 히스톤 옥타머 주위에 과도하게 비틀린 왼손잡이 DNA 감김이 나타났을 때,[57][58] 이 역설은 과학계에서 해결된 것으로 간주되었다.
Remove ads
같이 보기
- 핵산 시뮬레이션 소프트웨어 비교
- DNA 나노기술
- G-4중 나선
- DNA 분자 모델
- 핵산의 분자 구조: 디옥시리보핵산의 구조 (첫 번째 논문)
- Non-B 데이터베이스
- 삼중 나선 DNA
각주
Wikiwand - on
Seamless Wikipedia browsing. On steroids.
Remove ads
