O SARSr-CoV (do inglés Severe acute respiratory syndrome-related coronavirus ou coronavirus relacionado coa síndrome respiratoria aguda grave[nota 1] é unha especie de coronavirus que infecta os humanos, morcegos e algúns outros mamíferos.[2][3] É un virus con envoltura de ARN monocatenario de sentido positivo, que entra nas células hóspede uníndose ao receptor da superficie celular ACE2.[4] É un membro do xénero Betacoronavirus e do subxénero Sarbecoronavirus.[5][6]
- Este artigo trata dunha especie de coronavirus que comprende múltiples cepas. Para a cepa que causa a SARS, ver SARS-CoV. Para a cepa que causa a COVID-19, ver SARS-CoV-2.
| Coronavirus relacionado coa SARS[1] | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
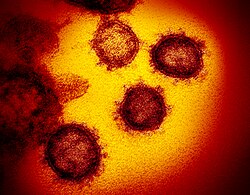 Micrografía electrónica de transmisión de virións de SARSr-CoV saíndo dunha célula hóspede cultivada en laboratorio. | |||||||||||||||
| Clasificación científica | |||||||||||||||
| |||||||||||||||
| Cepas | |||||||||||||||
|
SARS-CoV
| |||||||||||||||
Esta especie viral ten varias cepas. Dúas cepas deste virus causaron importantes gromos de enfermidades respiratorias graves en humanos: o SARS-CoV (ou SARS-CoV-1), que causou o gromo de 2002-2004 de síndrome respiratoria aguda grave (SARS), e o SARS-CoV-2, causante da pandemia por coronavirus de 2019–2021 da COVID-19.[7][8] Existen centos de cepas máis do SARSr-CoV, ningunha das cales infecta os humanos, senón outros mamíferos. Os morcegos son o seu principal reservorio, e varias cepas foron identificadas nas civetas Paradoxurus; ditas cepas probablemente son as antepasadas da cepa SARS-CoV.[7][9]
O coronavirus relacionado coa SARS foi un dos varios virus identificados pola Organización Mundial da Saúde (OMS) en 2016 como unha causa probable dunha epidemia futura nun novo plan desenvolvido despois da epidemia do virus Ébola para que se fixese unha investigación urxente e desenvolvementos sobre el antes e durante unha posible epidemia para conseguir tests de diagnóstico, vacinas e medicinas. A predición fíxose realidade co desencadeamento da pandemia por coronavirus de 2019–2021, que estendeu a COVID-19 por todo o mundo.[10][11]
Clasificación
O coronavirus relaconado coa SARS é membro do xénero Betacoronavirus (grupo 2) e do subxénero Sarbecovirus (subgrupo B).[12] Os sarbecovirus, a diferenza dos embecovirus ou alfacoronavirus, só teñen unha proteinase de tipo papaína (PLpro) en vez de dúas no marco de lectura aberto ORF1.[13] Determinouse que o SARSr-CoV era unha ramificación temperá dos betacoronavirus baseándose nun conxunto conservado de dominios que comparte con este grupo.[14][15]
Os morcegos son o principal hóspede reservorio dos coronavirus relacionados coa SARS. O virus coevolucionou nos morcegos reservorios durante un longo período de tempo.[16] Non foi ata tempos recentes que evolcionaron as cepas de coronavirus relacionados coa SARS e fixeron o salto entre especies desde os morcegos aos humanos, como no caso das cepas SARS-CoV e SARS-CoV-2.[4][17] Ambas as cepas descenderon dun único antepasado, pero fixeron o paso entre especies ata os humanos separadamente. O SARS-CoV-2 non é un descendente directo do SARS-CoV.[7] O 17 de marzo de 2020, os científicos informaron que o novo virus SARS-CoV-2 se orixinou de forma natural, e non doutro modo.[18][19]
Xenoma

O coronavirus relacionado coa SARS é un virus con envoltura de ARN monocatenario de sentido positivo. O seu xenoma é dunhas 30 kb, que é un dos maiores entre os virus de ARN. O virus ten 14 marcos de lectura abertos que se solapan nalgúns casos.[20] O xenoma ten unha caparuza 5′ metilada e unha cola 3′ poliadenilada.[21] Hai 265 nucleótidos na rexión non traducida 5' (5'UTR) e 342 nucleótidos na rexión non traducida 3' (3'UTR).[20]
A caparuza 5' metilada e a cola 3' poliadenilada permiten que o xenoma de ARN de sentido positivo sexa traducido directamente polos ribosomas da célula hóspede na entrada viral.[22] O SARSr-CoV é similar a outros coronavirus en canto ao comezo da expresión do seu xenoma, que se inicia coa tradución polos ribosomas da célula hóspede dos seus dous grandes marcos de lectura abertos iniciais (ORFs), 1a e 1b, que producen ambos poliproteínas.[20]
| Función das proteínas xenómicas (de orf1a a orf9b) do SARS-CoV | |
|---|---|
| Proteína | Función[23][24][25] |
| orf1a, orf1b |
Poliproteína replicase/transcriptase (pp1ab) (proteínas non estruturais) |
| orf2 | Proteína da espícula (S), adhesión e entrada do virus (proteína estrutural) |
| orf3a | Interacciona coas proteína estruturais S, E, M; Actividade de canle iónica; Regula á alza as citocinas e quimiocinas como a IL-8 e RANTES; Regula á alza o NF-κB e JNK; Induce a apoptose e parada do ciclo celular por medio das caspases 8 e 9, e por Bax, p53 e quinase MAP p38 |
| orf3b | Regula á alza citocinas e quimiocinas por medio de RUNX1b; Inhibe a produción de IFN de tipo I e sinalizando; Induce a apoptose e a parada do ciclo celular; |
| orf4 | Proteína da envoltura (E), ensamblaxe do virus e evaxinación (proteína estrutural) |
| orf5 | Proteína de membrana (M), ensamblaxe do virus e evaxinación (proteína estrutural) |
| orf6 | Potencia a síntese de ADN celular; Inhibesa produción e sinalización de IFN de tipo I |
| orf7a | Inhibe a síntese de proteínas celulares; Induce a resposta inflamatoria por NF-κB e promotor do IL-8; Regula á alza quimiocinas como o IL-8 e RANTES; Regula á alza a JNK e a quinase MAP p38; Induce a apoptose e a parada do ciclo celular |
| orf7b | Descoñecido |
| orf8a | Induce a apoptose pola vía mitocondrial |
| orf8b | Potencia a síntese de ADN celular |
| orf9a | Proteína da nucleocápside (N), empaquetamento do ARN viral (proteína estrutural) |
| orf9b | Induce a apoptose |
Coñécense as funcións de varias proteínas virais.[26] Os ORFs 1a e 1b codifican a poliproteína replicase/transcriptase, e os posteriores ORFs 2, 4, 5 e 9a codifican, respectivamente, as catro proteínas estruturais principais: espícula, envoltura, membrana e nucleocápside.[27] Os ORFs posteriores tamén codifican oito proteínas únicas (de orf3a a orf9b), coñecidas como proteínas accesorias, moitas sen ningún homólogo coñecido. As diferentes funcións das proteínas accesorias non se coñecen.[26]
Morfoloxía

A morfoloxía do coronavirus relacionado coa SARS é a característica da familia dos coronavirus. Estes virus son grandes partículas esféricas pleomórficas con proxeccións superficiais bulbosas que forman unha especie de coroa arredor das partículas víricas nas micrografías electrónicas.[28] O tamaño das partículas víricas está entre 80–90 nm. A envoltura do virus en micrografías electrónicas aparece como un definido par de cubertas densas aos electróns.[29]
A envoltura viral consta dunha bicapa lipídica na que están ancoradas as proteínas da membrana (M), envoltura (E) e espícula (S).[30] As proteínas da espicula orixinan as proxeccións bulbosas da superficie do virus. A interacción das proteínas da espícula co seu receptor da célula hóspede correspondente é fundamental para determinar o tropismo de tecido, a infectividade e o rango de especies ás que infecta o virus.[31][32]
Dentro da envoltura hai unha nucleocápside, que está formada por múltiples copias da proteína da nucleocápside (N), que están unidas ao ARN xenómico monocatanario de sentido positivo (~30 kb) nunha formación continua de tipo rosario de doas.[33][34] A envoltura de bicapa lipídica, as proteínas de membrana e a nucleocápside protexen o virus cando está fóra do hópede.[35]
Ciclo vital
O coronavirus relacionado coa SARS segue a estratexia de replicación típica dos coronavirus,[21][36][37][38][39] que se pode dividir nas seguintes fases:
Adhesión e entrada na célula

A unión do coronavirus relacionado coa SARS coa célula hóspede faise coa mediación da proteína da espícula e o seu receptor da superficie celular.[40] O dominio de unión ao receptor da proteína da espícula (RBD) recoñece e únese ao receptor, que é o enzima convertedor da anxiotensina 2 (ACE2).[4] Despois da adhesión, o virus pode entrar na célula hóspede por dúas vías diferentes. A vía que o virus tome depende da dispoñibilidade dunha protease do hóspede, que é a que corta e activa a proteína da espícula unida ao receptor.[41]
A primeira vía que pode usar o coronavirus da SARS para entrar na célula hóspede é por endocitose, na que o virus é captado nun endosoma. A proteína da espícula unida ao receptor é despois activada pola cisteina protease catepsina L dependente de pH do hóspede. A activación da proteína da espícula unida ao receptor causa un cambio conformacional e a subseguinte fusión da envoltura viral coa membrana do endosoma.[41]
Alternativamente, o virus pode entrar na célula hóspede directamente por clivaxe proteolítica da proteína da espícula unida ao receptor polas serina proteasess TMPRSS2 ou TMPRSS11D do hóspede na superficie celular.[42][43] No coronavirus da SARS, a activación da parte C-terminal da proteína da espícula desencadea a fusión da envoltura viral coa membrana da célula hóspede ao inducir cambios conformacionais que non se comprenden totalmente.[44]
Tradución do xenoma
| Función das proteínas non estruturais dos coronavirus (nsps)[45] | |
|---|---|
| Proteína | Función |
| nsp1 | Promove a degradación do ARNm do hóspede, bloquea a tradución do hóspede; bloquea a resposta inmunitaria innata |
| nsp2 | Únese ás proteínas prohibitinas; función descoñecida |
| nsp3 | Proteína transmembrana multidominio; interacciona coa proteína N; promove a expresión de citocinas; o dominio PLPro cliva a poliproteína pp1ab e bloque a resposta inmunitaria innata do hóspede; funcións descoñecidas doutos dominios |
| nsp4 | Proteína armazón transmembrana; permite adquirir a estrutura axeitada ás vesículas de dobre membrana (DMVs) |
| nsp5 | A 3CLPro cliva a poliproteína pp1ab |
| nsp6 | Proteína armazón transmembrana; función descoñecida |
| nsp7 | Forma complexos hexadecaméricos con nsp8; abrazadeira de procesividade para a RdRp (nsp12) |
| nsp8 | Forma un complexo hexadecamérico co nsp7; abrazadeira de procesividade para RdRp (nsp12); actúa como unha primase |
| nsp9 | Proteína de unión ao ARN (RBP) |
| nsp10 | Cofactores nsp16 e nsp14; forma un heterodímero con ambos; estimula a 2-O-MT (nsp16) e a actividade de ExoN (nsp14) |
| nsp11 | Función descoñecida |
| nsp12 | ARN polimerase ARN dependente (RdRp) |
| nsp13 | ARN helicase, 5′ trifosfatase |
| nsp14 | N7 Metltransferase, 3′-5′ exorribonuclease (ExoN); a N7 MTase engade a caparuza 5', ExoN fai a corrección de probas do xenoma |
| nsp15 | Endorribonuclease (NendoU) |
| nsp16 | 2′-O-Metiltransferase (2-O-MT); protexe o ARN viral de MDA5 |
Despois da fusión a nucleocápside pasa ao citoplasma, onde se libera o xenoma viral.[40] O xenoma actúa como un ARN mensaxeiro e os ribosomas da célula traducen dous terzos do xenoma, que se corresponden cos marcos de lectura abertos ORF1a e ORF1b, orixinando dúas grandes poliproteínas, chamadas pp1a e pp1ab.
A poliproteína máis grande pp1ab é o resultado dun cambio da pauta de lectura ribosómica -1 causado pola secuencia escorregante (UUUAAAC) e un pseudonó de ARN de augas abaixo no final do marco de lectura abero ORF1a.[46] O cambio de pauta de lectura ribosómica permite a tradución continua de ORF1a seguida da de ORF1b.[47]
As poliproteínas conteñen as súas propias proteases, PLpro e 3CLpro, que clivan as proteínas en sitios específicos diferentes. A clivaxe da poliproteína pp1ab orixina 16 proteínas non estruturais (de nsp1 a nsp16). As proteínas produto inclúen varias proteínas de replicación como unha ARN polimerase ARN dependente (RdRp), unha ARN helicase e unha exorribonuclease (ExoN).[38][47]
Replicación e transcrición

Varias proteínas non estruturais coalescen para formar un complexo multiproteico replicase-transcriptase (RTC).[47] A principal proteína replicase-transcriptase é a ARN polimerase ARN dependente (RdRp). Está directamente implicada na replicación e transcrición do ARN a partir dunha febra molde de ARN. As outras proteínas non estruturais do complexo axudan no proceso de replicación e transcrición.[45]
A proteína nsp15 é unha 3'-5' exorribonuclease que proporciona fidelidade extra ao proceso de replicación. A exorribonuclease proporciona unha función de corrección de probas ao complexo, do cal carece a ARN polimerase ARN dependente. De xeito similar, as proteínas nsp7 e nsp8 forman unha abrazadeira deslizante hexadecamérica que forma parte do complexo, que incrementa grandemente a procesividade da ARN polimerase ARN dependente.[45] Os coronavirus necesitan o incremento da fidelidade e da procesividade durante a síntese de ARN debido ao seu tamaño de xenoma relativamente grande en comparación con outros virus de ARN.[48]
Unha das principais funcións do complexo replicase-transcriptase é transcribir/replicar o xenoma viral. A RdRp é mediadora directa da síntese de moléculas de ARN subxenómicas de sentido negativo a partir do ARN xenómico positivo. Isto vai seguido da transcrición destas moléculas de ARN subxenómico de sentido negativo aos seus correspondentes ARNm de sentido positivo.[49]
O ARN xenómico de sentido positivo replicado convértee no xenoma da proxenie de virus. Os diversos ARNm máis pequenos son transcritos a partir do último terzo do xenoma do virus, que está a continución dos marcos de lectura ORF1a e ORF1b. Estes ARNm son traducidos ás catro proteínas estruturais (S, E, M e N) que formarán parte das partículas de virus da proxenie e tamén outras oito proteínas accesorias (de orf3 a orf9b) que axudan o virus.[50]
Ensamblaxe e liberación
A tradución do ARN ocorre denrro do retículo endoplasmático. As proteínas estruturais virais S, E e M móvense pola vía secretora no compartimento intermediario de Golgi. Alí, as proteínas M dirixen a maioría das interaccións proteína-proteína requirida para a ensamblaxe dos virus despois da súa unión á nucleocápside.[51]
Os virus da proxenie son liberados do hóspede por exocitose por medio de vesículas secretoras.[51]
Notas e referencias
Véxase tamén
Wikiwand in your browser!
Seamless Wikipedia browsing. On steroids.
Every time you click a link to Wikipedia, Wiktionary or Wikiquote in your browser's search results, it will show the modern Wikiwand interface.
Wikiwand extension is a five stars, simple, with minimum permission required to keep your browsing private, safe and transparent.
